개요
이전 포스팅에서는 서울시 코로나 확진자 현황 데이터를 크롤링 과정에 관한 내용을 소개했었습니다. 이번 포스팅은 본격적으로 파이썬(Python)의 다양한 데이터 분석 도구를 활용하여 EDA(Exploratory Data Analysis)를 수행한 과정을 작성하였습니다.
특별히, 이번 포스팅은 YouTube today오늘코드 채널에서 분석한 내용을 기반으로 작성하였으며, 개인적으로 2021년 2월 1일까지 수집된 서울시 코로나 확진자 현황 데이터로 확장하여 EDA를 수행하였습니다.
참고한 자료는 아래와 같습니다.
본문
필요한 모듈 및 데이터 로드
필요한 pandas, numpy 모듈을 import하고, 2021년 2월 1일까지 서울시 코로나 확진자 현황 데이터를 크롤링하여 .csv 파일을 불러와서 확인합니다.
- 총 24,395개 row와 7개 column으로 구성되어 있습니다.
- 데이터 수집 시점(2021년2월2일) 기준으로 최초 서울시 코로나 확진자는 2020년 1월 24일에 중국에 여행력을 가진 강서구 거주자이며, 가장 최근 서울시 코로나 확진자는 2021년 2월 1일에 발생한 마포구 거주자입니다.
import pandas as pd
import numpy as np
file_name = f'data/seoul-covid19-2021_02_01.csv'
df = pd.read_csv(file_name)
df = df.sort_values(by="연번", ascending=False)
df.head()
matplotlib를 이용해서 시각화를 수행할 때, 한글 폰트와 마이너스(-) 기호를 올바로 표시하기 위해 설정합니다.
import matplotlib.pyplot as plt
plt.rc("font", family="Malgun Gothic") # Windows
plt.rc("axes", unicode_minus=False)
# test
pd.Series([1,3,5,-7,9]).plot.bar(title="한글 제목")
3. 날짜별 확진자 분석
확진일과 확진자수의 관계를 분석하기 위해 먼저 확진일 칼럼 데이터의 전처리를 수행합니다. 확진일 칼럼 타입을 datetime으로 변경하고, 년, 월, 주로 분리합니다.
- 최초 확진자 발생일인 2020-01-24(1월 4주차)를 기준으로 주차별로 1씩 증가하며, 2021-01-03까지 53주차, 2021-01-04부터 1월 1주차로 적용됩니다.
df["확진일"] = pd.to_datetime(df["확진일"])
df["년"] = df["확진일"].dt.year
df["월"] = df["확진일"].dt.month
df["주"] = df["확진일"].dt.week
# test
df[["확진일", "년", "월", "주"]]
위 df의 전체 날짜를 기준으로 확진자 수 발생 추이를 선 그래프로 시각화합니다. df.plot()은 기본적으로 데이터를 선 그래프로 표현하고, plt.axhline()은 분포의 특징을 더 쉽게 알아볼 수 있도록 x축과 평행한 라인을 출력합니다.
- 2020년 8월 이전까지 크진 않지만, 서울시에서 확진자는 꾸준하게 발생했습니다.
- 2020년 8월 이후에 확진자가 눈에 띄게 증가했습니다.
- 2020년 12월 이후에는 확진자가 이전보다 훨씬 급증하여 600명에 이르는 확진자가 발생한 날도 확인할 수 있습니다.
df["확진일"].value_counts().sort_index().plot(figsize=(15,4))
plt.axhline(200, color='red', linestyle=":")
전체 데이터에서 확진자가 가장 많이 발생한 날을 확인합니다.
- 2020년 12월 17일에 서울시 확진자가 588명으로 가장 많이 발생했습니다.
day_count = df["확진일"].value_counts().sort_index()
day_count[day_count == day_count.max()]
day_count
2020년 12월 17일의 접촉력 데이터 빈도수를 확인했습니다.
- 동부구치소 관련 확진자와 기타 확진자 접촉(n차 감염)에 의한 확진자가 가장 많았고, 데이터 수집 시점(2020년 2월 2일) 기준으로 아직 감염경로가 불분명한 확진자도 다수 존재합니다.
df[df["확진일"]=='2020-12-17']["접촉력"].value_counts().sort_values().plot.barh()
'월'별 확진자수
2020년과 2021년을 분리하여 월별 확진자 수 변화 추이를 시각화했습니다.
- 위에서 살펴봤듯이, 2020년에는 8월, 11월, 12월에 서울시 코로나 확진자가 증가하며, 2021년에는 작년 12월에 이어 코로나 확진자가 여전히 발생하는 추세입니다.
df.groupby(["월", "년"])["연번"].count().unstack().plot.bar(figsize=(10,4))
'주'별 확진자수
2020년과 2021년을 분리하여 주별 확진자수 변화 추이를 시각화했습니다.
- 위 '월'별 확진자수와 동일한 추이를 보입니다.

4. 각 날짜별 확진자수를 저장한 데이터프레임 생성
각 날짜별 발생한 확진자수만 저장한 새로운 데이터프레임을 생성합니다. 이때, 확진자가 발생하지 않은 날짜에는 Nan으로 채웁니다.
1. 최초 확진자 발생 날짜(2020-01-24) ~ 데이터 수집 시점을 기준으로 가장 최근 확진자 발생날짜(2020-02-01)까지 모든 날짜를 저장한 데이터프레임을 생성합니다.
- 총 375일을 확인할 수 있습니다.
first_day = df.iloc[-1, 2]
last_day = df.iloc[0, 2]
days = pd.date_range(first_day, last_day)
df_days = pd.DataFrame({"확진일자": days})
df_days.head()
2. 확진자 발생날짜 별 확진자수를 기존 df에서 추출합니다.
- 확진자가 발생한 날은 총 343일입니다.
daily_case = df["확진일"].value_counts()
df_daily_case = daily_case.to_frame()
df_daily_case.columns = ["확진수"]
df_daily_case
3. 1, 2에서 생성한 데이터프레임을 병합(merge) 합니다.
- 전체 375일 중에 확진자가 발생하지 않은 날은 확진수 칼럼에 Nan으로 저장되었습니다.
all_days = df_days.merge(df_daily_case, left_on="확진일자", right_index=True, how='left')
all_days
5. 누적 확진자 수 계산
위에서 생성한 데이터프레임을 가지고 누적 확진수를 계산하고, 선 그래프로 시각화합니다.
- 2020년 8월 이전까지 누적 확진자가 천천히 증가하며, 이후로 증가폭이 다소 상승합니다.
- 2020년 12월 이후로 누적 확진자의 증가폭이 매우 가파르게 상승합니다.
all_days["확진수"] = all_days["확진수"].fillna(0)
all_days["누적확진"] = all_days["확진수"].cumsum()
cum_day = all_days.set_index("확진일자")
# 시각화
cum_day.plot(figsize=(15,4))
plt.grid(True)

누적 확진자수와 확진자수의 차이가 크기 때문에 로그 스케일로 변환하여 분석합니다.
- '로그 스케일'은 상대적으로 작은 값은 큰 값으로 스케일링하고, 큰 값은 작은 값으로 스케일링하는 특성이 있기 때문에 작은 값의 분포를 좀 더 자세히 확인할 수 있습니다.
- 확진자가 없는 날의 '0'은 로그 스케일로 변환 시, (-) 무한대로 발산하므로, 아래와 같이 라인이 중간에 잘려 있습니다. 하지만, 대다수 날짜에 확진자가 발생했음을 알 수 있습니다.
np.log(cum_day["확진수"]).plot(figsize=(15,4))
np.log(cum_day["누적확진"]).plot()
plt.grid(True)
6. '월', '요일' 별 확진자 분석
'월'과 '요일' 별 확진자 수를 분석하기 위해 데이터를 2020년, 2021년으로 분리하고, '월'을 index로 하고, '요일'을 column으로 하는 테이블을 생성합니다.
# 2020년
all_days_2000= all_days[all_days["확진일자"]==2020]
all_days_2000_week = all_days_2000.groupby(["확진월", "확진요일"])["확진수"].sum()
all_days_2000_week = all_days_2000_week.unstack()
all_days_2000_week.columns = ["월", "화", "수", "목", "금", "토", "일"]
# 2021년
all_days_2001 = all_days[all_days["확진일자"]==2021]
all_days_2001_week = all_days_2001.groupby(["확진월", "확진요일"])["확진수"].sum()
all_days_2001_week = all_days_2001_week.unstack()
all_days_2001_week.columns = ["월", "화", "수", "목", "금", "토", "일"]
먼저, 2020년 월별, 요일별 확진자 분포를 구별하기 쉽게 df.style을 적용합니다.
- 2020년 8월부터 모든 요일에 확진자가 급증했다가 잠잠해졌으며, 11월부터 다시 확진자가 급증하는 것을 확인할 수 있습니다.
all_day_2020_week.style.background_gradient(cmap="Blues")
- 각 월별로 평일에 발생한 확진자가 주말에 발생한 확진자보다 많음을 알 수 있습니다. 이는 평일 대비 주말에 코로나 검사 횟수가 적기 때문인 것으로 추정됩니다.
all_day_2020_week.style.highlight_max(axis=1)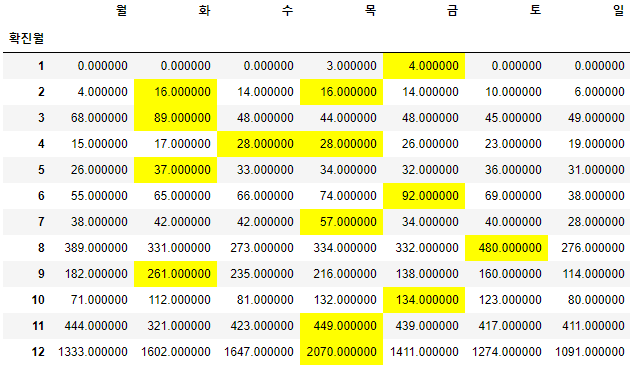
다음으로, 2021년 데이터에도 동일하게 df.style을 적용합니다.
- 2021년 1월은 전년도 12월에 이어 모든 요일에 대하여 많은 확진자가 발생했습니다.
- 2021년 2월은 2월 1일 하루치 일부 데이터만 수집된 형태이므로, 큰 의미를 부여하기 어렵습니다.
all_day_2020_week.style.background_gradient(cmap="Greens")
- 2021년 1월은 전년도와 다르게 평일 대비 주말에 확진자가 더 많이 발생했음을 알 수 있습니다.
all_day_2021_week.style.highlight_max(axis=1)
7. 거주지별 확진자 분석
먼저, 거주지 데이터를 확인해보면, 거주지명 앞뒤로 공백이 들어가 있는 경우를 확인할 수 있습니다.
df["거주지"].unique()
앞뒤 공백을 모두 제거하는 전처리 수행 후, 공백이 제거된 것을 확인할 수 있습니다.
df["거주지"] = df["거주지"].str.strip()
df["거주지"].unique()
이제 전체 거주지 별 확진자 분포를 시각화합니다.
- '기타' 및 '타시도'는 거주지가 서울이 아니지만, 서울에서 확진 판정을 받은 환자입니다.
- '기타'로 분류된 곳에 거주하는 확진자가 가장 많으며, 서울 이외에 '타시도'에 거주하는 확진자도 적지 않음을 알 수 있습니다. 만약 이 환자들이 무증상이었더라면, 더 넓은 지역 사회로 번질 가능성도 있었음을 추론할 수 있습니다.
gu_etc_count = df["거주지"].value_counts()
gu_etc_count.sort_values().plot.barh(figsize=(10,12))
8. 접촉력 별 확진자 분석
먼저, 접촉력 데이터에서 아래와 같이 수정이 필요한 데이터를 확인할 수 있었고, 전처리를 수행합니다.
- "성동구 소재 병원 관련(\\\\\\'21.1월)" 및 "성동구 소재 병원 관련(\\\\\\\\\\\\\\'21.1월)"을 "성동구 소재 병원 관련('21.1월)"로 수정
- '기타확진자 접촉'을 '기타 확진자 접촉'으로 수정
df.loc[(df["접촉력"].str.contains("성동구 소재 병원 관련"))&(df["접촉력"].str.contains("21.1월")), "접촉력"] = "성동구 소재 병원 관련('21.1월)"
df.loc[df["접촉력"].str.contains("기타확진자 접촉"), "접촉력"] = '기타 확진자 접촉'
이제 상위 30개 접촉력 별 확진자 수를 시각화합니다.
- 기타 확진자 접촉이 가장 많은 것으로 보아 지역사회 n차 감염이 가장 큰 확산세의 원인인 것을 알 수 있습니다.
- 감염경로 조사 중인 경우가 뒤를 따르는데, 이는 역학 조사의 한계가 있음을 유추해 볼 수 있습니다.
- 위 두 가지 문제에 대한 해결책이 필요한 시점임을 알 수 있습니다.
contact_count_top = df["접촉력"].value_counts().sort_values().tail(30)
contact_count_top.plot.barh(figsize=(10,12))
(월, 접촉력) 별 확진자 분석
접촉력에 따른 확진자를 더 세밀하게 분석하기 위해 확진자 수가 많은 상위 15개 접촉력에 대하여 2020년, 2021년으로 분리하고, 각 월마다 접촉력에 따른 확진자 분포를 시각화했습니다.
top_group = df[df["접촉력"].isin(contact_count_top.tail(15).index)]
2020년
# 2020년
top_group_2020 = top_group[top_group["년"]==2020].groupby(["접촉력", "월"])["연번"].count().unstack().fillna(0).astype(int)
top_group_2020.style.background_gradient(cmap="Blues")- 1월에 최초 '해외유입' 확진자에 의한 n차 감염이 2월로 이어지면서 점차 감염 속도가 증가했습니다.
- 3월부터는 '해외유입'과 'Ace 손해보험' 확진자가 급증하면서 데이터 수집 시점(2021-02-02)까지도 결국 감염경로를 파악하지 못한 환자도 급증했습니다. 또한, '해외유입' 확진자도 증가했습니다.
- 4월에는 여전히 '해외유입' 확진자가 많았으나 지역사회 감염이 다소 안정기에 접어든 것을 확인할 수 있습니다.
- 5월에는 갑작스레 '이태원 클럽 관련' 확진자가 폭증했습니다.
- 6월과 7월에 n차 감염이 확산되었고, 감염경로가 불투명한 확진자도 다수 발생했습니다.
- 8월에는 '8.15서울도심집회' 관련 확진자와 '성북구 사랑제일교회' 관련 확진자가 폭증했으며, n차 감염과 감염경로가 불투명한 확진자도 다수 발생했습니다.
- 9월, 10월에는 계속해서 n차 감염과 감염경로가 불투명한 확진자가 다수 발생했습니다.
- 11월에는 '강서구 댄스교습관련 시설' 확진자와 '마포구 홍대새교회 관련' 확진자, '서초구 사우나 관련' 확진자, '해외 유입' 확진자도 증가해였고, n차 감염과 감염경로가 불투명한 확진자도 폭증했습니다.
- 12월에는 '동부구치소' 관련 확진자와 '종로구 소재 파고다타운 관련' 확진자가 폭증하였고, n차 감염과 감염경로가 불투명한 확진자가 2020년도에 가장 크게 증가하였습니다.

2021년
top_group_2021 = top_group[top_group["년"]==2021].groupby(["접촉력", "월"])["연번"].count().unstack().fillna(0).astype(int)
top_group_2021.style.background_gradient(cmap="Greens")- 1월에는 전년도 12월에 이어 '동부구치소 관련' 확진자와 '해외유입' 확진자가 발생했으며, n차 감염과 감염경로가 불투명한 확진자도 매우 크게 발생했습니다.
- 2월은 2월 1일의 일부 데이터만 존재하기 때문에 큰 의미가 없습니다.

'감염경로 조사 중'인 확진자 분석
감염경로가 불투명한 확진자 추세를 시각화하고, 전체 확진자와 비교하기 위해 시각화했습니다.
- '감염경로가 불투명한 확진자'는 2020년 8월에 증가 후에 점차 감소했습니다.
- 이후 2020년 11월에 재차 증가하고, 2020년 12월에는 폭증하는 것을 확인할 수 있습니다.
- 이후 2021년 1월에는 다소 감소하는 경향을 확인할 수 있습니다.
df_unknown = df[df["접촉력"]=='감염경로 조사중']
unknown_weekly_case = df_unknown.groupby(["년", "월"])["연번"].count()
unknown_weekly_case.plot.bar(figsize=(15,4), rot=45)
이제 전체 확진자 추세와 비교하기 위해 '년 - 주 - 전체 확진수' 데이터프레임과 '년 - 주 - 불명확 진수' 데이터프레임을 생성합니다.
# 1) 전체 확진수 데이터프레임
all_weekly_case = df[["년", "주"]].value_counts().to_frame()
all_weekly_case.columns = ["전체확진수"]
# 2) 감염경로가 불명확한 확진수 데이터프레임
unknown_weekly_case = df_unknown[["년", "주"]].value_counts().to_frame()
unknown_weekly_case.columns = ["불명확진수"]
생성한 2개의 데이터프레임을 병합(merge)합니다. 이때, merge() 함수의 how='inner'인데, 추후 불명확 진수가 발생한 날짜만 시각화를 수행하기 위함입니다.
unknown_case = all_weekly_case.merge(unknown_weekly_case, left_index=True, right_index=True)
unknown_case = unknown_case.sort_index()
unknown_case
unknown_case.plot(figsize=(15,4))
plt.grid(True)- 감염경로가 불투명한 확진자는 주로 전체 확진자수와 비슷한 경향을 띄고 있음을 알 수 있습니다.
- 확진자수가 증가함에 따라 감염경로를 조사할 수 있는 역학 조사 인력 부족이나 올바른 이동 동선을 제공하지 않는 문제 등에도 대응이 필요할 것이라 예측할 수 있습니다.

마지막으로, 전체 확수 중에 불명 확진수 비율을 시각화했습니다.
- 전반적으로 감염경로가 불명한 확진자 비율이 크고 작음을 반복하다가 2020년 12월 이후에는 그 비율이 지속해서 큰 상태임을 확인할 수 있습니다.
unknown_case["확인중비율"] = (unknown_case["불명확진수"] / unknown_case["전체확진수"]) * 100
unknown_case["확인중비율"].plot.bar(figsize=(15,4)
plt.grid(True)
9. 퇴원, 사망 여부 분석
데이터 수집 시점(2021-0202)에서 확진자의 퇴원, 사망 비율을 분석했습니다. 퇴원현황 칼럼에는 'Nan', '사망', '퇴원' 3가지 데이터가 존재합니다. 2개의 새로운 칼럼(퇴원, 사망)을 생성하여 True, False를 저장하되 'Nan'은 False로 저장합니다.
df["퇴원"] = df["퇴원현황"].str.contains("퇴원", na=False)
df["사망"] = df["퇴원현황"].str.contains("사망", na=False)
퇴원, 사망 비율을 각각 확인합니다.
- 퇴원 비율: 약 84%
- 사망 비율: 약 1%
- 대체로 순조롭게 퇴원하고 있으며, 사망 확률도 매우 낮음을 알 수 있습니다.
df["퇴원"].value_counts(normalize=True)
df["사망"].value_counts(normalize=True)
가장 오래 입원 중인 서울 거주 환자
이번에는 서울에 거주하는 확진자 중, 가장 오랫동안 입원중인 확진자를 분석합니다.
- 데이터 수집 시점(2021-02-02)을 기준으로 '강남구 소재 대우디오빌플러스'에 접촉하여 2020년 9월 15일에 확진 판정을 받은 마포구 거주 환자가 가장 오랜 기간 입원 중이었습니다.
df_seoul = df[df["거주지"].isin(gu_count.index] # 서울에 거주하는 환자 데이터
df_seoul[(df_seoul["퇴원"]==False) & (df_seoul["사망"]==False)].tail()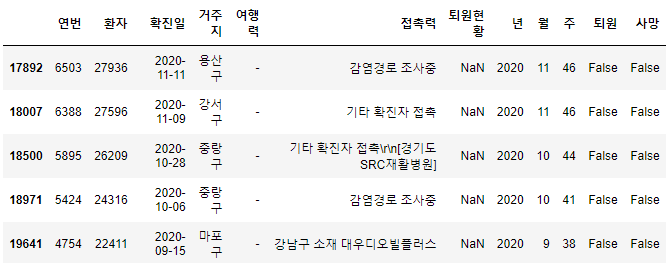
10. 여행력 별 확진자 분석
먼저, 여행력 칼럼 데이터에 대하여 몇 가지 전처리를 수행했습니다.
여행력이 없는 경우, '-'가 저장되어 있는데, 이를 Nan으로 변경합니다. 또한, 환자 번호가 잘못 들어간 경우가 있는데, 이를 올바른 여행력 데이터를 추측하기 어렵다는 판단하에 row 데이터를 제거합니다.
# 전처리를 수행할 때, 새로운 칼럼을 생성한 후에 전처리 하는 것이 좋음
df["해외"] = df["여행력"]
df["해외"] = df["해외"].replace('-', np.nan) # - 를 Nan으로 교체
# df_oversea: 해외 칼럼 데이터를 가진 경우만 필터링
df_oversea = df[df["해외"].notnull()].copy()
df_oversea.drop(df_oversea[df_oversea["해외"]=='28238'].index, inplace=True) # 환자 번호가 잘못 들어간 row 데이터 삭제
df_oversea.drop(df_oversea[df_oversea["해외"]=='21263'].index, inplace=True) # 환자 번호가 잘못 들어간 row 데이터 삭제
국가명 그룹핑 수행
해외 칼럼에 국가의 개수가 많기 때문에 동일한 대륙에 속한 국가를 그룹핑하고, 동일한 국가를 다른 이름으로 표기한 데이터를 동일한 국가명으로 수정합니다. 다만, '유럽'과 '남미'에 속하는 나라에 여행력을 가진 경우에는 어떻게 처리하는 게 맞는지 추가 고민이 필요할 것 같습니다.
- 유럽 국가 분류
- 남미 국가 분류
- 각 중국, UAE, 필리핀, 미국으로 국가명 통일
europe = "체코,헝가리,오스트리아,이탈리아,프랑스,모로코,독일,스페인,영국,폴란드,터키,아일랜드"
europe = europe.replace(",", "|")
south_america = "브라질,아르헨티아,칠레,볼리비아,멕시코,페루"
south_america = south_america.replace(",", "|")
df_oversea.loc[df_oversea["해외"].str.contains(europe), "해외"] = '유럽'
df_oversea.loc[df_oversea["해외"].str.contains(south_america), "해외"] = '남미'
df_oversea.loc[df_oversea["해외"].str.contains('중국|우한'), "해외"] = '중국'
df_oversea.loc[df_oversea["해외"].str.contains('아랍에미리트'), "해외"] = 'UAE'
df_oversea.loc[df_oversea["해외"].str.contains('필리핀'), "해외"] = '필리핀'
df_oversea.loc[df_oversea["해외"].str.contains('미국'), "해외"] = '미국'
df_oversea["해외"]
확진일자와 여행력 국가별 확진자 분석
먼저, 여행력을 가진 확진자 데이터프레임에서 확진일 및 해외 칼럼 별 확진자 수를 계산합니다.
day_oversea = df_oversea.groupby(["확진일", "해외"])["연번"].count()
day_oversea
이렇게 생성된 MultiIndex 데이터프레임의 해외 칼럼의 각 국가별 누적 확진자 수를 계산합니다. 이를 위해 MultiIndex 데이터프레임의 해외 칼럼을 각 국가별로 그룹핑을 수행해야 합니다. df.groupby()의 level=[1]로 저장하면, 가장 최상위 인덱스("확진일")의 하위 인덱스("해외")를 그룹핑 기준으로 삼을 수 있으며, level=[-1]로 지정해도 동일한 결과를 얻을 수 있습니다.
day_oversea_cumsum = day_oversea.groupby(level=[1]).cumsum()
day_oversea_cumsum
이제 누적 확진자가 가장 자주 발생한 국가를 분석하여 시각화합니다.
- 미국, 유럽을 여행력으로 가진 확진자가 가장 자주 발생했음을 알 수 있습니다.
df_day_oversea_cumsum = df_day_oversea_cumsum.reset_index()
df_day_oversea_cumsum = df_day_oversea_cumsum.rename(columns={"연번": "누적확진수"})
oversea_count = df_day_oversea_cumsum["해외"].value_counts()
oversea_count.sort_values().plot.bar(figsize=(10,12))
plt.grid(True)
다음으로 국가별 누적 확진자가 가장 많은 상위 10개 국가의 확진일에 따른 누적 확진자 수 변화를 분석합니다.
- 미국, 유럽, 남미, 필리핀, 러시아 순으로 누적 확진자가 가장 많음을 알 수 있습니다.
df_day_oversea_top_10 = df_day_oversea_cumsum.groupby("해외")["누적확진수"].max().sort_values(ascending=False)[:10]
df_day_oversea_top_10 = df_day_oversea_top_10.index
df_day_oversea_top_10
- 유럽을 여행력으로 가진 확진자가 2020년 1월에 가장 먼저 발생한 것으로 확인됩니다.
- 시간이 흐를수록 미국을 여행력으로 가진 누적 확진수가 증가하며 유럽을 여행력으로 가진 누적 확진자수가 뒤를 따릅니다.
df_day_oversea_cumsum = df_day_oversea_cumsum[df_day_oversea_cumsum["해외"].isin(df_day_oversea_top_10)]
df_day_oversea_cumsum = df_day_oversea_cumsum.set_index("확진일")
df_day_oversea_cumsum.pivot(columns="해외").plot(figsize=(24,4))
누적 확진자수가 가장 많은 '미국'을 선 그래프로 시각화합니다.
- 2020년 3월 이후로 누적 확진수가 크게 한 번 증가합니다.
- 이후로 원만하게 증가하다가 12월 이후로 급증합니다.
df_day_oversea_cumsum[df_day_oversea_cumsum["해외"]=='미국']]["누적확진수"].plot()
거주지와 여행력 국가별 확진자 분석
- 여행력을 가진 확진자는 강남구에 가장 많이 거주하고 있으며, 기타 지역, 용산구, 마포구 순으로 많이 거주하고 있습니다.
oversea_count_gu = df_oversea["거주지"].value_counts()
oversea_count_gu.sort_values().plot.barh(figsize=(10,12))
거주지 별 전체 확진자 수 및 여행력을 가진 확진자 수 분석
거주지 별 전체 확진자 데이터프레임 및 여행력을 가진 확진자 데이터프레임을 생성하고, 병합(merge)하여 한 개의 데이터프레임으로 생성합니다.
# 거주지 별 전체 확진자
all_count_gu = df["거주지"].value_counts()
df_all_gu = all_count_gu.to_frame()
df_all_gu.columns = ["전체확진수"]
# 거주지 별 여행력을 가진 확진자
oversea_count_gu = df_oversea["거주지"].value_counts()
df_oversea_gu = oversea_count_gu.to_frame()
df_oversea_gu.columns = ["여행력존재확진수"]
위에서 생성한 데이터프레임을 병합니다.
df_all_oversea_case = df_all_gu.merge(df_oversea_gu, left_index=True, right_index=True)
df_all_oversea_case
병합한 데이터프레임을 시각화하고, 일부 거주지별 전체 확진수 중에 여행력을 가진 확진수 비율을 계산합니다.
df_all_oversea_case.sort_values(by="여행력존재확진수").plot.barh(figsize=(15,8))
- 전체 확진자 중에 여행력이 존재하는 확진자 비율은 용산구, 강남구, 마포구, 서초구 순으로 많습니다.
df_all_oversea_case["비율"] = (df_all_oversea_case["여행력존재확진수"] / df_all_oversea_case["전체확진수"])*100
df_all_oversea_case.sort_values(by="비율", ascending=False)
여행력을 가진 확진자의 거주지 별 퇴원 여부
여행력을 가진 확진자의 거주지 별 퇴원 빈도를 시각화합니다.
- 강남구에 거주하는 확진자가 가장 많이 퇴원했음을 확인할 수 있습니다.
oversea_finish_count = df_oversea.groupby(["거주지". "퇴원"])["연번"].count().unstack()
oversea_finish_count = oversea_finish_count.fillna(0).astype(int)
oversea_finish_count.plot.bar(rot=30, figsize=(15,4))
plt.grid(True)
하지만 각 거주지마다 전체 확진자수가 다르기 때문에 퇴원비율을 계산하는 것이 좀 더 명확한 수치라 생각합니다. 퇴원비율이 작은 거주지 순서대로 정렬하여 일부 데이터만 확인합니다.
- 거주지와 상관없이 확진자의 퇴원비율은 대부분 높음을 알 수 있습니다.
- 다만, 중구에 거주하는 확진자의 퇴원비율은 약 66.7%입니다.
- 2020년에 제공되었던 서울시 코로나 확진자 현황 데이터는 환자가 입원한 서울시 지정 병원을 확인할 수 있었는데, 대체로 거주지와 가까운 병원에 입원함을 할 수 있었습니다. -> 이를 토대로 서울시 중구에는 치료 센터가 상대적으로 적은 가능성을 유추할 수 있습니다.
oversea_finish_count_prop = pd.DatFrame(oversea_finish_count[:,True] / (oversea_finish_count[:,True] + oversea_finish_count[:,False]))
oversea_finish_count_prop = oversea_finish_count_prop.rename(columns={0: "퇴원비율"}).sort_values(by="퇴원비율")
oversea_finish_count_prop
여행력을 가진 확진자의 월별 발생 빈도
2020년, 2021년 데이터를 분리하여 여행력을 가진 확진자가 월별로 발생한 빈도수를 시각화합니다.
- 2020년 3월부터는 여행력을 가진 확진자수가 급증한 이후에 감소세를 보입니다.
- 2020년 9월 이후로 마지막 데이터 수집 시점까지 계속해서 증가세를 보입니다. (2021년 2월은 2월 1일 하루치 일부 데이터만 수집된 형태이므로, 큰 의미를 부여하기 어렵습니다.)
df_oversea.groupby(["월", "년"])["연번"].count().unstack().plot()
여행력을 가진 확진자의 월별, 거주지별 발생 빈도
2020년, 2021년 데이터를 분리하고, 동일한 결과를 총 3가지 방법(gropby, crosstab, pivot_table)으로 시각화합니다.
1) groupby
- 2020년
- 여행력을 가진 확진자가 증가하는 3월에 발생한 확진자의 거주지는 강남구, 서초구, 송파구 순으로 많이 거주하고 있습니다.
- 9월 이후에 발생하는 확진자의 거주지는 기타 지역, 용산구, 마포구에서 수치가 높은 경우가 있었으며, 대부분은 다양한 거주지를 가지고 있음을 알 수 있습니다.
# 2020년
month_gu_2020 = df_oversea[df_oversea["년"]=='2020'].groupby(["월", "거주지"])["연번"].count().unstack()
month_gu_2020 = month_gu_2020.fillna(0).astype(int)
month_gu_2020.style.background_gradient(cmap="Blues")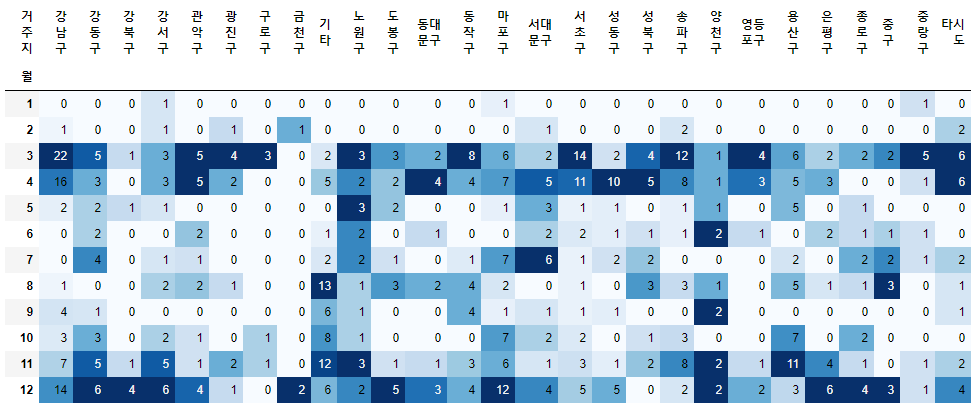
- 2021년
- 2021년 1월에는 여행력을 가진 확진자의 거주지가 용산구, 강남구, 성북구 순으로 많으며, 다양한 지역에 거주하고 있음을 확인할 수 있습니다.
- 2021년 2월은 2월 1일 하루치 일부 데이터만 수집된 형태이므로, 큰 의미를 부여하기 어렵습니다.
# 2021년
month_gu_2021 = df_oversea[df_oversea["년"]=='2021'].groupby(["월", "거주지"])["연번"].count().unstack()
month_gu_2021 = month_gu_2021.fillna(0).astype(int)
month_gu_2021.style.background_gradient(cmap='Greens')
2) crosstab
위와 동일한 시각화 결과를 Pandas corsstab을 이용하여 출력합니다. 2020년 데이터만 예시로 시각화를 수행했습니다.
month_gu_2020 = pd.crosstab(df_oversea[df_oversea["년"]==2020]["월"], df_oversea["거주지"])
month_gu_2020.style.bar()
3) pivot_table
위와 동일한 시각화 결과를 Pandas pivot_table을 이용하여 출력합니다. 2020년 데이터만 예시로 시각화를 수행했습니다.
month_gu_2020 = pd.pivot_table(df_oversea[df_oversea["년"]==2020], values="연번", index="월", columns="거주지", aggfunc='count', fill_value=0)
month_gu_2020
여행력을 가진 확진자의 거주지별 국가 분석
2020년, 2021년 데이터를 분리하고, 각 연도별 여행력이 많은 국가 10개를 거주지별로 분석합니다. 여기서는 gropby, pivot_table로 시각화했으며, 2020년만 예시로 수행했습니다.
1) groupby
- 미국, 유럽을 여행력으로 가진 확진자가 가장 많으며, 전반적으로 다양한 지역에 거주하는 것을 알 수 있습니다.
df_oversea_2020 = df_oversea[df_oversea["년"]==2020]
top_country_2020 = df_oversea_2020["해외"].value_counts()[:10].index
group_oversea_gu_2020 = df_oversea_2020[df_oversea_2020["해외"].isin(top_country_2020)].groupby(["해외", "거주지"])["연번"].count().unstack()
group_oversea_gu_2020.fillna(0).astype(int).style.background_gradient("Blues")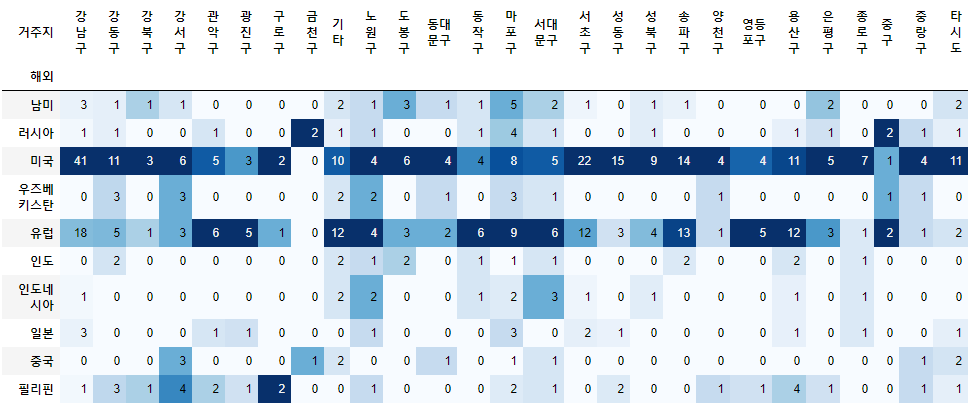
2) pivot_table
group_oversea_gu_2020 = pd.pivot_table(df_oversea_2020[df_oversea_2020["해외"].isin(top_country_2020)], index="해외", columns="거주지", values="연번", aggfunc='count', fill_value=0)
group_oversea_gu_2020.style.background_gradient(cmap="Blues")
마무리
지금까지 서울시에서 제공하는 코로나 발생 현황 데이터에 대한 EDA 수행 과정에 대하여 말씀드렸습니다. EDA 절차를 하나의 그림으로 표현하면 아래와 같습니다.
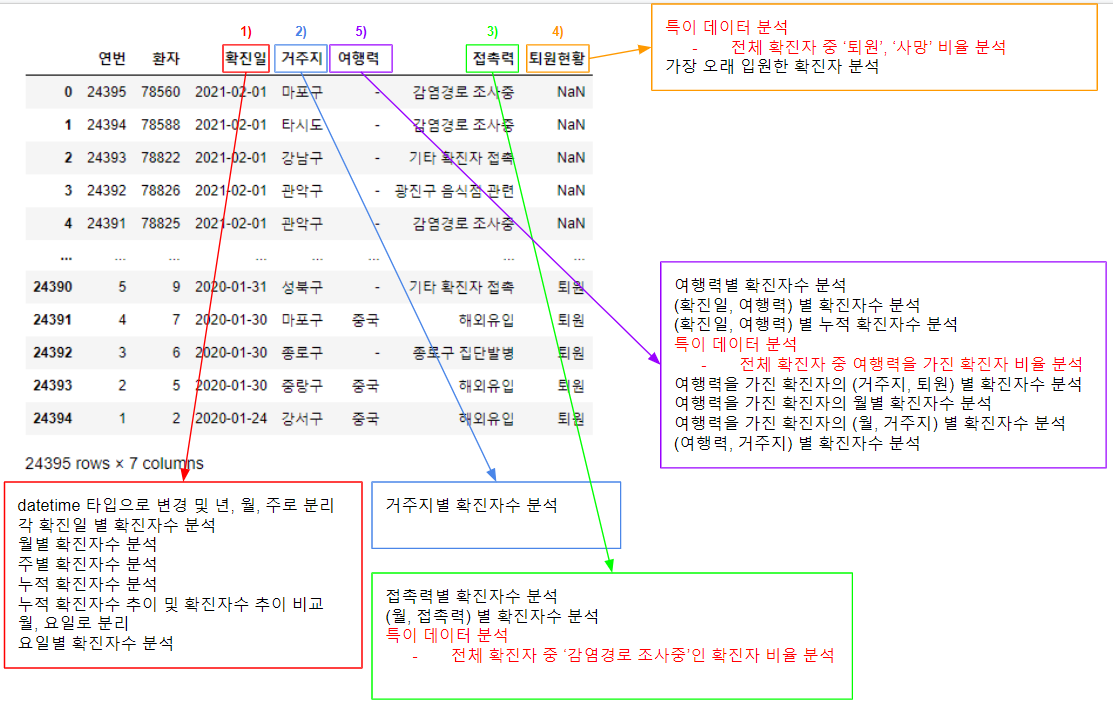
데이터 분석을 위한 인사이트를 얻는 데 도움이 되셨으면 좋겠습니다 :)
'데이터 분석 > Python' 카테고리의 다른 글
| 파이썬 서울시 코로나19 현황 분석하기 - 1. 데이터 크롤링 (0) | 2021.02.09 |
|---|
